---
tags: tutorial, 生醫, T3-bio
---
# 國網中心GP1生醫核心設施 - 台灣杉三號生醫專用節點使用說明
## 申請加入GP1核心設施服務計畫
* 若您尚未申請GP1核心設施計算服務,請參考[申請服務流程](https://lions.nchc.org.tw/ngsApplyDoc.jsp)。
* 若您已經申請計算服務,但尚未加入核心設施iService服務計畫,請參考[申請加入說明](https://man.twcc.ai/@nchcbio/SJg8LWQEC)。
## 登入台灣杉三號生醫專用節點
* 生醫專用節點主機位置:t3-c4.nchc.org.tw 或 203.145.216.54。
* 請勿使用台灣杉1號進行計算,如此會衍生出額外的費用,請使用者配合。
* 亦請勿使用登入節點主機的CPU與GPU進行計算,以免影響登入節點運作與排程系統效能,敬請配合。
* 帳號密碼:iservice內 **會員中心->會員資訊->主機帳號資訊** ,可以找到您的帳號或修改密碼
* One Time Password(OTP)請參閱:https://man.twcc.ai/@TWCC-III-manual/SJwbCxzqO#%E5%8F%96%E5%BE%97-OTP-%E8%AA%8D%E8%AD%89%E7%A2%BC
## 資料上傳
* 請使用 t3-c4.nchc.org.tw 節點上傳資料,可以使用sftp、scp、rsync上傳資料。
### SFTP
請用下面指令登入sftp
```
sftp youruserid@t3-c4.nchc.org.tw
```
出現下面訊息後,輸入登入方法、主機密碼、OTP
```
Connecting to t3-c4.nchc.org.tw...
Please select the 2FA login method.
1. Mobile APP OTP
2. Mobile APP PUSH
3. Email OTP
Login method: 1 # 1 or 2 or 3
Password: # 主機密碼
OTP: XXXXXX # OPT
```
成功後出現
```
sftp>
```
## 生醫計算環境節點與費率
|節點種類|節點數量|單一節點核心數|記憶體大小|國科會計畫費率|一般學界費率|
|:--:|:--:|:--:|:--:|:--:|:--:|
|CPU節點|45|56|384G|0.08元/核心小時|0.24元/核心小時|
|GPU節點|2|8GPU(V100)|768G|10元/核心小時|25元/核心小時|
|2TB大記憶體節點|1|36|2TB|0.56元/核心小時|1.68元/核心小時|
|4TB大記憶體節點|1|72|4TB|0.56元/核心小時|1.68元/核心小時|
|6TB大記憶體節點|2|112|6TB|0.56元/核心小時|1.68元/核心小時|
<font color="#f00">※ **請注意:GPU與大記憶體節點費率較高,使用時請謹慎選擇排程。**</font>
## 高速計算儲存空間
目前使用者在以下位置所擁有的空間如下:
| 空間 | 容量 |
|:-------------------------- |:------------------:|
| /home/$USER | 100G |
| /staging/biology/$USER | 5T |
| /work/$USER | 1500GB |
| /staging/reserve/PI_folder | 付費空間需另外申請 |
| 巨量資料儲存服務 | 付費空間需另外申請 |
※ 爲鼓勵國科會計畫用戶使用本中心此最新的超級電腦,自2021年9月1日起,調高國科會計畫每個帳號之高速檔案系統(HFS)儲存空間的暫存工作目錄區域(/work)免費額度,由原本的100 GB,提高到1500 GB。而家目錄區域(/home)免費額度則維持不變,仍為100 GB。
※ 國科會計畫之帳號於新建立時,系統將預設免費儲存空間額度為家目錄區域100 GB,暫存工作目錄為100 GB。若有需要提高暫存工作區域之免費額度,須請客戶自行調高。用戶欲調整免費儲存空間之額度,請由申購高速儲存系統(HFS)介面進行調整,相關說明文件可參考下面台灣杉三號可用儲存資源與調整之說明文件連結。
※ 台灣杉三號可用儲存資源與調整之說明文件請參考: https://man.twcc.ai/@TWCC-III-manual/HyOgKIiuu
<font color="#f00">※ **請注意:調整額度時,請勿超過1500GB,避免被收取超用額度的費用,超過的額度收費依據國網中心HFS公告收費,並將由使用者負擔,每GB每月定價4元,一個月1TB收費4000元。**</font>
## 巨量資料儲存空間
* 巨量資料儲存空間[使用說明請參閱連結](https://man.twcc.ai/@nchcbio/Hki4S0eMh)。
## 儲存空間管理政策與使用須知
1. 高速計算磁碟空間(/work/\$USER, /staging/biology/\$USER)為計算工作短期暫存之用,若需長期保留,請將檔案移至核心設施[巨量資料儲存服務](https://man.twcc.ai/@Ldk_QYrOR2yo3m8Cb1549A/rkegDKslF#%E5%B7%A8%E9%87%8F%E8%B3%87%E6%96%99%E5%84%B2%E5%AD%98%E7%A9%BA%E9%96%93 "巨量資料儲存服務")。
2. 高速計算磁碟空間(/work/\$USER, /staging/biology/\$USER)預設權限為700(rwx------),若需要分享給實驗室夥伴,請參閱[資料權限設定、共享](https://man.twcc.ai/@Ldk_QYrOR2yo3m8Cb1549A/rkegDKslF#2-%E8%B3%87%E6%96%99%E6%AC%8A%E9%99%90%E8%A8%AD%E5%AE%9A%E3%80%81%E5%85%B1%E4%BA%AB "資料權限設定")章節,請使用者妥善管理上述空間的權限,本核心設施無法代為管理,若造成檔案外洩、遺失或刪除,本核心設施不負任何還原或賠償責任。
3. [巨量資料儲存服務](https://man.twcc.ai/@Ldk_QYrOR2yo3m8Cb1549A/rkegDKslF#%E5%B7%A8%E9%87%8F%E8%B3%87%E6%96%99%E5%84%B2%E5%AD%98%E7%A9%BA%E9%96%93 "巨量資料儲存服務")提供您實驗室產出資料的備份,採用Erasure Code(4+2)保護機制來保護您的資料,可用性及完整性高,不另外進行資料備份。
5. 本核心設施不會對高速計算磁碟空間做任何備份,若系統毀損或檔案刪除皆無法復原。
6. 磁碟空間上的檔案用戶需自行備份保留,若發生檔案毀損、遺失或刪除,本核心設施不負任何還原或賠償責任。
7. 本儲存空間可讓您上傳、儲存、下載內容,您的資料仍屬於您所有。對於您的任何內容 (包括您在儲存空間中上傳、共用或儲存的所有資訊和檔案),我們都不會聲明擁有權。因此,如果您決定與他人共用文件,我們可以提供這項功能。您可以跟他人共同編輯檔案,但內容的「擁有者」為控制內容和其用途的使用者。
8. 儲存服務裡的共用設定可讓您控管其他人如何使用您儲存空間中的檔案。檔案的隱私權設定則是取決於檔案所在的資料夾或儲存空間。只要您還沒指定共用對象,您儲存空間中的檔案就只有您本人能夠存取。您可以與他人共用自己的檔案,也可以將內容的管理權轉移給其他使用者。如果您在他人與您共用的資料夾或儲存空間中建立或放置檔案,這些檔案將可能會沿用所在資料夾或儲存空間的共用設定,也可能會沿用擁有權設定,我們不會與他人共用您的檔案和資料。
9. 網際網路資料的傳輸不能保證百分之百的安全,本核心設施服務將努力保護您的資料安全,使用加密機制進行資料傳輸,高速計算儲存服務可透過ssh連線傳輸資料;巨量資料儲存服務可使用S3通訊協定的ssl加密連線或是IBM Aspera(Aspera CLI指令ascp或是Aspera Desktop Client)進行加密傳輸,來確保您資料傳輸時是受到保護的。
10. 本服務不會要求您提供特殊或具敏感性的個人資料,例如醫療或健康紀錄、政治或宗教信仰、性取向、性生活、犯罪紀錄或基因,也請您不要提供此類訊息予我們。
11. 本服務採用適當之安全措施,防止您的資料被竊取、竄改、洩露或毀損,包括對資料的儲存、處理和安全措施,以及實體上的安全措施進行內部檢查,以防杜我們儲存資料的系統遭受入侵。
12. 當您使用儲存時發現有可能危害本中心營運作業、資訊安全之資通安全事件時,請與本核心設施服務人員聯繫。(生醫小組客服 電話: 03-5776085#370 電子郵件: bio.nchc@niar.org.tw)
## 公用軟體安裝位置與模組使用指令
### 1. 公用軟體安裝位置
軟體絕對路徑在 /opt/ohpc/Taiwania3/pkg/biology/軟體名稱/軟體版本 內:
`/opt/ohpc/Taiwania3/pkg/biology`
<font color="#f00">※ *舊版公用軟體會留存供需要的使用者使用,如果因使用舊版軟體所造成的資安漏洞風險,請自行負責。*</font>
### 2. 如何使用模組載入程式?
部分軟體有寫成模組,可自動匯入軟體所需環境,使用方法如下:
* 列出所有biology可用模組:
```
module load biology
module avail
```
* 加載模組:
```
module load <module_name>
```
例如:
a. 如要加載 R/4.3.3、BWA/0.7.17:
```
module load biology
module load R/4.3.3
module load BWA/0.7.17
```
b. 如要加載 gcc/12.3.0
```
module load biology
module load gcc/12.3.0
```
* 卸載模組:
```
module unload gcc/12.3.0
```
* 列出已加載的模組:
```
module list
```
* 顯示模組信息:
```
module show R/4.3.3
```

### 3. 如何自行安裝 R package
* 載入 R,並執行 R
```
module load biology R/4.2.1
R
```
* 使用 .libPaths()指令 來確認載入package的位置。 預設只有T3軟體資料夾,如下
```
> .libPaths()
[1] "/work/opt/ohpc/Taiwania3/pkg/biology/R/R-4.2.1/library"
```
* 此時安裝packages時,會詢問是否使用個人的位置來安裝,均回答yes
```
> install.packages('ggplot2')
Warning in install.packages("ggplot2") :
'lib = "/work/opt/ohpc/Taiwania3/pkg/biology/R/R-4.2.1/library"' is not writable
Would you like to use a personal library instead? (yes/No/cancel) yes
Would you like to create a personal library
‘/home/u00cwh00/R/x86_64-pc-linux-gnu-library/4.2’
to install packages into? (yes/No/cancel) yes
```
* 安裝成功後會以後優先載入個人位置內的package。 使用.libPaths()指令查詢如下
```
> .libPaths()
[1] "/home/u00cwh00/R/x86_64-pc-linux-gnu-library/4.2"
[2] "/work/opt/ohpc/Taiwania3/pkg/biology/R/R-4.2.1/library"
```
* 可用以下指令查詢 所有package所在的所有位置,會以載入優先順序列出(以ggplot2為例)
```
> installed_packages <- as.data.frame(installed.packages()[, c("Package", "LibPath")])
> installed_packages[installed_packages$Package == "ggplot2", ]
Package LibPath
ggplot2 ggplot2 /home/u00cwh00/R/x86_64-pc-linux-gnu-library/4.2
ggplot2.1 ggplot2 /work/opt/ohpc/Taiwania3/pkg/biology/R/R-4.2.1/library
```
* 如果要使用的Library path,不是第一順位的話,請加 lib.loc="libpath" 來載入,如下
```
> library(ggplot2, lib.loc = "/work/opt/ohpc/Taiwania3/pkg/biology/R/R-4.2.1/library")
```
## Partition(Queue) 資源設定
|Partition|記憶體配置(`--mem`)|核心數搭配(`-c`)|時間限制|個人Job上限|使用節點|
|:--:|:--:|:--:|:--:|:--:|:--:|
|ngsTest|7G|1|10 mins|2|CPU節點|
|ngsConsole|7G|1|unlimit|2|CPU節點|
|ngs7G|7G|1|48 hrs|1000|CPU節點|
|ngs13G|13G|2|48 hrs|500|CPU節點|
|ngs26G|26G|4|96 hrs|250|CPU節點|
|ngs53G|53G|8|96 hrs|120|CPU節點|
|ngs92G|92G|14|unlimit|80|CPU節點|
|ngs186G|186G|28|unlimit|40|CPU節點|
|ngs372G|372G|56|unlimit|20|CPU節點|
|ngs224core|184G*4(sbatch 不用指定mem)|56*4 (sbatch 參數使用: -c 56 -n 4)|unlimit|4|CPU節點|
|ngs448core|184G*8(sbatch 不用指定mem)|56*8 (sbatch 參數使用: -c 56 -n 8)|unlimit|2|CPU節點|
|ngs512G|500G|9|unlimit|8|2,4TB大記憶體節點|
|ngs1T_18|1000G|18|unlimit|4|2,4TB大記憶體節點|
|ngs2T_36|2000G|36|unlimit|2|2,4TB大記憶體節點|
|ngs4T_72|4000G|72|unlimit|1|4TB大記憶體節點|
|ngs2T_28|2000G|28|unlimit|6|6TB大記憶體節點|
|ngs3T_56|3000G|56|unlimit|4|6TB大記憶體節點|
|ngs6T_112|6000G|112|unlimit|2|6TB大記憶體節點|
|ngs1gpu|90G|6CPU/1GPU(sbatch 參數使用: -c 6 --gres=gpu:1)|unlimit|4|GPU節點|
|ngs2gpu|180G|12CPU/2GPU (sbatch 參數使用: -c 12 --gres=gpu:2)|unlimit|2|GPU節點|
|ngs4gpu|360G|24CPU/4GPU (sbatch 參數使用: -c 24 --gres=gpu:4)|unlimit|1|GPU節點|
|ngs8gpu|720G|48CPU/8GPU (sbatch 參數使用: -c 48 --gres=gpu:8)|unlimit|1|GPU節點|
|ngs1gput|90G|6CPU/1GPU (sbatch 參數使用: -c 6 --gres=gpu:1)|48hr|8|GPU節點|
|ngs2gput|180G|12CPU/2GPU (sbatch 參數使用: -c 12 --gres=gpu:2)|48hr|4|GPU節點|
|ngs4gput|360G|24CPU/4GPU (sbatch 參數使用: -c 24 --gres=gpu:4)|24hr|2|GPU節點|
|ngs8gput|720G|48CPU/8GPU (sbatch 參數使用: -c 48 --gres=gpu:8)|24hr|1|GPU節點|
|ngscourse|13G|2|48 hrs|500|CPU節點|
|ngscourse92G|92G|14|unlimit|80|CPU節點|
※ 為確保記憶體與核心數能有效且充分利用,因此參數設定必須符合上述排程與記憶體核心數搭配與限制。
## 工作指令稿
### 1. 使用SLURM排程系統來送出工作
**請編寫一個檔案內容如下:**
* SLURM Job Script (CPU Queue)範例如下:
```shell
#!/usr/bin/sh
#SBATCH -A MST109178 # Account name/project number
#SBATCH -J Job_name # Job name
#SBATCH -p ngs53G # Partition Name 等同PBS裡面的 -q Queue name
#SBATCH -c 8 # 使用的core數 請參考Queue資源設定
#SBATCH --mem=53g # 使用的記憶體量 請參考Queue資源設定
#SBATCH -o out.log # Path to the standard output file
#SBATCH -e err.log # Path to the standard error ouput file
#SBATCH --mail-user=XXXX@niar.org.tw # email
#SBATCH --mail-type=BEGIN,END # 指定送出email時機 可為NONE, BEGIN, END, FAIL, REQUEUE, ALL
echo 'Hello world!' ##這邊寫入你要執行的指令
##以BWA為例
module load biology
module load BWA/0.7.17
bwa mem
```
※ 上面的core數與記憶體搭配,請[參考Queue資源設定或排程限制](https://man.twcc.ai/@Ldk_QYrOR2yo3m8Cb1549A/rkegDKslF#PartitionQueue-%E8%B3%87%E6%BA%90%E8%A8%AD%E5%AE%9A),要設對工作才能正常送出,請注意。
* SLURM Job Script (GPU Queue)範例如下:
```shell
#!/usr/bin/sh
#SBATCH -A MST109178 # Account name/project number
#SBATCH -J Job_name # Job name
#SBATCH -p ngs1gpu # Partition Name 等同PBS裡面的 -q Queue name
#SBATCH -c 6 # 使用的core數 請參考Queue資源設定
#SBATCH --mem=90g # 使用的記憶體量 請參考Queue資源設定
#SBATCH --gres=gpu:1 # 使用的GPU數 請參考Queue資源設定
#SBATCH -o out.log # Path to the standard output file
#SBATCH -e err.log # Path to the standard error ouput file
#SBATCH --mail-user=XXXX@niar.org.tw # email
#SBATCH --mail-type=BEGIN,END # 指定送出email時機 可為NONE, BEGIN, END, FAIL, REQUEUE, ALL
nvidia-smi ##這邊寫入你要執行的指令
```
* **寫完工作指令稿(jobscript.sh)後送出執行**
執行指令:`sbatch jobscript.sh`
執行結果:
```shell=
[USER@lgn304 ~]$ sbatch jobscript.sh
Submitted batch job 84684
```
進行工作遞交後,將會獲得一個 Job ID (上述範例為84684)。
### 2.送工作時指定參數
將上述script改為如下指令,並在最後加入job.sh即可。
```wrap
sbatch -A MST109178 -J Job_name -p ngs48G -c 14 --mem=46g -o out.log -e err.log --mail-user=XXXX@niar.org.tw --mail-type=BEGIN,END job.sh
```
如果要執行的工作只有一行指令,可以利用 --wrap="" 參數傳入指令,無需再寫script
```wrap
sbatch -A MST109178 -J Job_name -p ngs48G -c 14 --mem=46g -o out.log -e err.log --mail-user=XXXX@niar.org.tw --mail-type=BEGIN,END --wrap="ls /opt/ohpc/Taiwania3/pkg/biology"
```
### 3.引入外部變數
如果有變數要引入Job中使用,可以使用 --export= 來指定參數
```
A=5
b='test'
sbatch --export=A=$A,b=$b job.sh
```
### 4.Array Job
若要使用 Array Job 可在Jobscript內使用 --array 定義 Index 範圍
,在Task內部可使用 SLURM_ARRAY_TASK_ID 這個環境變數讀取該Task的Index。
**範例:**
```shell
#!/usr/bin/sh
#SBATCH -A MST109178 # Account name/project number
#SBATCH -J Job_name # Job name
#SBATCH -p ngs53G # Partition Name 等同PBS裡面的 -q Queue name
#SBATCH -c 8 # 使用的core數 請參考Queue資源設定
#SBATCH --mem=53g # 使用的記憶體量 請參考Queue資源設定
#SBATCH --array=1-10 # 定義index範圍為1~10
echo $SLURM_ARRAY_TASK_ID # SLURM_ARRAY_TASK_ID 為該Task的index
```
其他Array Job詳細用法請見:
https://slurm.schedmd.com/job_array.html
## 查看job狀態/刪除工作
### 1. 列出送出的工作
指令:`squeue -u [username]`
範例:

### 2. 個別工作詳細資訊
指令:`scontrol show job [job ID]`
範例:

### 3. 刪除工作
指令:`scancel -i [job ID]`
## 進階
### 1. 查詢使用空間的用量、限制
#### a. 查詢work、home 空間
指令:`/usr/lpp/mmfs/bin/mmlsquota -u $USER --block-size auto fs01 fs02`

** $USER請輸入您的主機帳號
** fs01是/work
** fs02是/home
#### b.查詢 /staging/biology 空間
指令:`/usr/lpp/mmfs/bin/mmlsquota -u $USER --block-size auto 5Kstaging:biology`

** $USER請輸入您的主機帳號
** 5Kstaging:biology為/staging/biology/$USER
#### c. 查詢 /staging/reserve 空間
指令:`/usr/lpp/mmfs/bin/mmlsquota -j $Fileset --block-size auto 5Kstaging`

** $Fileset請輸入您的在/staging/reserve/$Fileset目錄名稱
### 2. 資料權限設定、共享
#### a. 設定 /staging/reserve/$Fileset 空間權限
Fileset擁有者,可進行目錄空間與檔案分享,設定權限請注意需完成下列兩行指令。
#### b. 分享$Fileset讀取權限給someone帳號
指令:`setfacl -R -m u:someone:r-X,g::---,o::--- /staging/reserve/$Fileset`
#### c. 設定讀取繼承權限給someone帳號,未來該目錄內檔案繼承目錄權限,自動設定權限給someone帳號。
指令:`setfacl -d -m u:someone:r-X,g::---,o::--- /staging/reserve/$Fileset`
** 若您需要讓someone有權限寫入分享目錄,請將r-X改為rwX即可。
#### d. someone要有分享的資料夾上層所有的資料夾d的x權限才能被使用
如果要一次開啟上層所有資料夾的x權限可以使用下列方式
```=sh
# Replace /path/to/folder with the actual folder path
folder="/path/to/folder"
# Replace username with someone's user name
someone="username"
# Loop through all parent directories and set the execute permission for the user
while [ "$folder" != "/" ]; do
setfacl -m u:${someone}:x "$folder"
folder=$(dirname "$folder")
done
```
設定完成後,可以透過指令getfacl進行確認。
```shell
[USER@lgn304 ~]$ getfacl /staging/reserve/$Fileset
# file: Fileset
# owner: yourself
# group: root
user::rwx
user:someone:rwx
group::---
mask::rwx
other::---
default:user::rwx
default:user:someone:rwx
default:group::---
default:mask::rwx
default:other::---
```
移除someone帳號的權限,與設定時一樣需執行兩行指令,移除即時讀取與繼承權限。
指令:`setfacl -x u:someone /staging/reserve/$Fileset`
指令:`setfacl -d -x u:someone /staging/reserve/$Fileset`
### 3. 查詢工作記憶體使用狀況
指令:`sacct -j $jobid -o JobID,JobName,Partition,User,NCPUS,AllocNodes,maxrss,Start,End,Elapsed,State`
```
JobID JobName Partition User NCPUS AllocNodes MaxRSS Start End Elapsed State
------------ ---------- ---------- --------- ---------- ---------- ---------- ------------------- ------------------- ---------- ----------
152151 GATK30X ngs192G u00cwh00 56 1 2021-09-14T18:58:46 2021-09-15T07:33:25 12:34:39 COMPLETED
152151.batch batch 56 1 116474744K 2021-09-14T18:58:46 2021-09-15T07:33:25 12:34:39 COMPLETED
152151.exte+ extern 56 1 1204K 2021-09-14T18:58:46 2021-09-15T07:33:25 12:34:39 COMPLETED
```
$jobid.batch行 MaxRSS欄位 顯示數字即為記憶體用量最大值。
### 4. 查詢歷史工作
指令:`sacct --starttime YYYY-MM-DD -u $USER -o JobID,JobName,Partition,State,ExitCode`
```
JobID JobName Partition State ExitCode
------------ ---------- ---------- ---------- --------
3592069 _norwayte+ ngs48G TIMEOUT 0:0
3592069.bat+ batch CANCELLED 0:15
3592069.ext+ extern COMPLETED 0:0
3608109 _norwayte+ ngs48G COMPLETED 0:0
3608109.bat+ batch COMPLETED 0:0
3608109.ext+ extern COMPLETED 0:0
3846029 _norwayte+ ngs92G RUNNING 0:0
3846029.bat+ batch RUNNING 0:0
3846029.ext+ extern RUNNING 0:0
```
### 5. 查詢可用排程與節點資源
#### a. 查詢可用排程狀態
指令: `qstat -ngs`
```shell
Total Total Running Running Pending Pending
Queue_Name STAT Job Core Job Core Job Core
---------------------------------------------------------------
ngsTest up 4 4 0 0 4 4
ngs7G up 2 2 0 0 2 2
ngs12G up 1 1 0 0 1 1
ngs24G up 1 1 0 0 1 1
ngs48G up 59 826 59 826 0 0
ngs96G up 0 0 0 0 0 0
ngs192G up 0 0 0 0 0 0
ngs224core up 0 0 0 0 0 0
ngs448core up 0 0 0 0 0 0
ngs512G up 0 0 0 0 0 0
ngs1T_18 up 0 0 0 0 0 0
ngs2T_36 up 0 0 0 0 0 0
ngs4T_72 up 0 0 0 0 0 0
ngs2T_28 up 0 0 0 0 0 0
ngs3T_56 up 0 0 0 0 0 0
ngs6T_112 up 0 0 0 0 0 0
ngs1gpu up 0 0 0 0 0 0
ngs2gpu up 0 0 0 0 0 0
ngs4gpu up 0 0 0 0 0 0
ngs8gpu up 0 0 0 0 0 0
---------------------------------------------------------------
SUM 67 834 59 826 8 8
```
#### b. 查詢排程與節點狀態
指令: `sinfo -s|grep ngs`
```shell
ngsTest up 10:00 23/22/0/45 cpn[3856-3900]
ngs7G up 2-00:00:00 23/22/0/45 cpn[3856-3900]
ngsConsole up infinite 23/22/0/45 cpn[3856-3900]
ngs13G up 2-00:00:00 23/22/0/45 cpn[3856-3900]
ngs26G up 4-00:00:00 23/22/0/45 cpn[3856-3900]
ngs53G up 4-00:00:00 23/22/0/45 cpn[3856-3900]
ngs92G up infinite 23/22/0/45 cpn[3856-3900]
ngs186G up infinite 23/22/0/45 cpn[3856-3900]
ngs372G up infinite 23/22/0/45 cpn[3856-3900]
ngs224core up infinite 23/22/0/45 cpn[3856-3900]
ngs448core up infinite 23/22/0/45 cpn[3856-3900]
ngs512G up infinite 0/2/0/2 bgm[3005-3006]
ngs1T_18 up infinite 0/2/0/2 bgm[3005-3006]
ngs2T_36 up infinite 0/2/0/2 bgm[3005-3006]
ngs4T_72 up infinite 0/1/0/1 bgm3005
ngs2T_28 up infinite 0/2/0/2 bgm[3001-3002]
ngs3T_56 up infinite 0/2/0/2 bgm[3001-3002]
ngs6T_112 up infinite 0/2/0/2 bgm[3001-3002]
ngs1gpu up infinite 1/1/0/2 gpn[3001-3002]
ngs2gpu up infinite 1/1/0/2 gpn[3001-3002]
ngs4gpu up infinite 1/1/0/2 gpn[3001-3002]
ngs8gpu up infinite 1/1/0/2 gpn[3001-3002]
```
#### c. 查詢排程限制
指令: `sacctmgr show qos -o format=name,MinTRES%28,MaxTRES%28,MaxJobsPU|grep ngs`
```shell
Name MinTRES MaxTRES MaxJobsPU
---------- ---------------------------- ---------------------------- ---------
ngstest cpu=1,mem=7G cpu=1,mem=7G 2
ngs7g cpu=1,mem=7G cpu=1,mem=7G 1000
ngs224core cpu=224 cpu=224 4
ngs448core cpu=448 cpu=448 2
ngs512g cpu=9,mem=500G cpu=9,mem=500G 8
ngs1t18 cpu=18,mem=1000G cpu=18,mem=1000G 4
ngs2t36 cpu=36,mem=2000G cpu=36,mem=2000G 2
ngs4t72 cpu=72,mem=4000G cpu=72,mem=4000G 1
ngs2t28 cpu=28,mem=2000G cpu=28,mem=2000G 6
ngs3t56 cpu=56,mem=3000G cpu=56,mem=3000G 4
ngs6t112 cpu=112,mem=6000G cpu=112,mem=6000G 2
ngs1gpu cpu=6,gres/gpu=1,mem=90G cpu=6,gres/gpu=1,mem=90G 16
ngs2gpu cpu=12,gres/gpu=2,mem=180G cpu=12,gres/gpu=2,mem=180G 8
ngs4gpu cpu=24,gres/gpu=4,mem=360G cpu=24,gres/gpu=4,mem=360G 4
ngs8gpu cpu=48,gres/gpu=8,mem=720G cpu=48,gres/gpu=8,mem=720G 2
ngsconsole cpu=1,mem=7G cpu=1,mem=7G 2
ngs13g cpu=2,mem=13G cpu=2,mem=13G 500
ngs26g cpu=4,mem=26G cpu=4,mem=26G 250
ngs53g cpu=8,mem=53G cpu=8,mem=53G 120
ngs92g cpu=14,mem=92G cpu=14,mem=92G 80
ngs186g cpu=28,mem=186G cpu=28,mem=186G 60
ngs372g cpu=56,mem=372G cpu=56,mem=372G 30
```
使用指令: `scontrol show partition ngs12G` 來了解ngs12G partition的QoS
```shell
PartitionName=ngs12G
AllowGroups=MST109178 AllowAccounts=mst109178 AllowQos=ALL
AllocNodes=ALL Default=NO QoS=ngs12g
DefaultTime=NONE DisableRootJobs=NO ExclusiveUser=NO GraceTime=0 Hidden=NO
MaxNodes=UNLIMITED MaxTime=2-00:00:00 MinNodes=0 LLN=NO MaxCPUsPerNode=UNLIMITED
Nodes=cpn[3856-3894]
PriorityJobFactor=1 PriorityTier=100 RootOnly=NO ReqResv=NO OverSubscribe=NO
OverTimeLimit=NONE PreemptMode=OFF
State=UP TotalCPUs=2184 TotalNodes=39 SelectTypeParameters=NONE
JobDefaults=(null)
DefMemPerNode=UNLIMITED MaxMemPerNode=UNLIMITED
```
#### d. 查詢節點使用狀態
指令: `ngsnodes`
```shell
Nodes Status for NGS and Protein Platform
Node T_ncpus U_ncpus A_ncpus T_ngpus U_ngpus A_ngpus T_Mem U_Mem A_Mem nJobs State Partition Used
cpn3871 56 55 1 0 0 0 187G 183G 4G 2 busy ngs48G,ct2k
cpn3872 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct2k
cpn3873 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3874 56 54 2 0 0 0 187G 180G 7G 2 busy ngs48G,ct560
cpn3875 56 42 14 0 0 0 187G 143G 45G 4 free ngs48G,ngs48G,ngs7G,ct2k
cpn3876 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3877 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3878 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct560
cpn3879 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct560
cpn3880 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3881 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct560
cpn3882 56 40 16 0 0 0 187G 136G 52G 3 free ngs48G,ngs48G,ct2k
cpn3883 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3884 56 40 16 0 0 0 187G 136G 52G 3 free ngs48G,ngs48G,ct2k
cpn3885 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3886 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct2k
cpn3887 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3888 56 40 16 0 0 0 187G 136G 52G 3 free ngs48G,ngs48G,ct2k
cpn3889 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3890 56 55 1 0 0 0 187G 185G 2G 4 busy ngs48G,ngs48G,ct2k,ct2k
cpn3891 56 55 1 0 0 0 187G 185G 2G 4 busy ngs48G,ngs48G,ct2k,ct2k
cpn3892 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3893 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct2k
cpn3894 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3895 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3896 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3897 56 55 1 0 0 0 187G 185G 2G 4 busy ngs48G,ngs48G,ct2k,ct2k
cpn3898 56 55 1 0 0 0 187G 183G 4G 3 busy ngs48G,ct2k,ct2k
cpn3899 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
cpn3900 56 54 2 0 0 0 187G 184G 4G 4 busy ngs48G,ngs48G,ngs48G,ct2k
bgm3001 112 0 112 0 0 0 6046G 0G 6046G 0 free
bgm3002 112 0 112 0 0 0 6046G 0G 6046G 0 free
gpn3001 56 0 56 8 0 8 187G 0G 187G 0 free
T = Total ; U = Used ; A = Available
```
** 由於台灣杉3號有NRQ(Non-Reserved Queue)設計,當GP1核心設施生醫專用資源閒置時,將會釋放至NRQ供其他用戶使用,若GP1核心設施使用者送出工作後,NRQ使用者的工作會被中斷,優先計算GP1核心設施使用者所送出之工作。
** 查詢節點狀態時,請注意Partition Used欄位,若有非ngs開頭Partition使用生醫專用節點,代表生醫專用資源有多餘資源可供使用,並非資源被占滿。
### 6. 查詢是否已加入生醫核心設施計畫
指令: `get_su_balance`
```shell
[USER@lgn304 ~]$ get_su_balance
{"PROJECT_ID":"MST109178","PROJECT_NAME":"國家生醫數位資料與分析運算雲端服務平台III","SU_BALANCE":"237010.1157"}
```
**您必須要加入MST109178計畫才能使用生醫專用計算環境。
### 7. 檔案加密方式
#### a.高速計算儲存空間檔案加密
##### 壓縮並加密 指令:
```
tar -czvf - your_file_or_your_directory | openssl aes-256-cbc -pbkdf2 -salt -k password -e -out /path/to/file.tar.gz
```
##### 解密並解壓縮 指令:
```
openssl aes-256-cbc -pbkdf2 -salt -k password -d -in /path/to/file.tar.gz | tar xzf -
```
#### b.巨量資料儲存空間檔案加密
可使用IBM Aspera(Aspera CLI指令ascp或是Aspera Desktop Client)來加解密上傳後的檔案,相關使用說明請參考 [Client-Side Encryption-at-Rest (EAR)](https://www.ibm.com/docs/en/ahts/3.9.6?topic=ascp-client-side-encryption-rest-ear)
## 桌面環境、圖形環境
桌面環境/圖形環境,提供使用者視覺化資料的環境,例如使用IGV、Rstudio 等圖形化界面的程式。
*** 注意如果為計算類工作請使用排程系統送到後端節點計算,以免影響其他使用者。
### Thinlinc桌面環境(for windows and mac)
1. 下載Thinlinc, 安裝後打開。
https://www.cendio.com/thinlinc/download/
2. 填入hostname以及主機帳號,密碼欄位鍵入1或2或3來選擇OTP驗證方式(1:APP OTP,2:APP PUSH,3:email OTP)

3. 點選connect後,在跳出的視窗內輸入主機密碼

4. 選Forward

5. 選擇桌面,GNOME或Xfce均可使用。

6. GNOME桌面點選左上角Activity,再點選左邊選單符號,即可看到terminal程式

7. 進入terminal後可以執行有圖形化界面的程式,例如:
* IGV: /opt/ohpc/Taiwania3/pkg/biology/IGV/IGV_v2.10.3/igv.sh
* Rstudio: /opt/ohpc/Taiwania3/pkg/biology/RSTUDIO/rstudio_v2021.09.0-351/bin/rstudio
### Mobaxterm(for Windows)
1. 下載 MobaXterm
https://mobaxterm.mobatek.net/download.html
2. 安裝並開啟
3. 開啟session,或編輯已經建立的session

4. 填入主機以及帳號, Advanced SSH setting 裡面 X-11 Fowarding 記得打勾

5. 進入執行有圖形化界面的程式,例如:
* IGV: /opt/ohpc/Taiwania3/pkg/biology/IGV/IGV_v2.10.3/igv.sh
6. 即可跳出程式視窗

### XQuartz(for Mac)
1. 下載 Quartz
https://www.xquartz.org/
2. 安裝並開啟 XQuartz

4. Xquartz terminal內輸入 ssh -Y username@t3-c4.nchc.org.tw

5. 進入後執行有圖形化界面的程式,例如:
* IGV: /opt/ohpc/Taiwania3/pkg/biology/IGV/IGV_v2.10.3/igv.sh
6. 即可跳出程式視窗

## 軟體使用說明
### SRAtoolkit
1.第一次使用請先使用以下指令進入界面設定:
```
/opt/ohpc/Taiwania3/pkg/biology/SRAToolkit/sratoolkit_v2.11.1/bin/vdb-config --interactive
```
2.進入界面後,按s 儲存,按x離開。
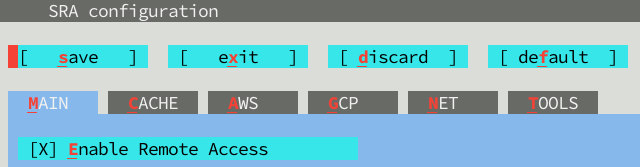
3.設定完畢後即可使用。
### 如何跑 R code
1. 先編輯一個檔案內容為 R code,假設名字為 myRcode.r,以下為檔案模擬內容:
```r=
#!/usr/bin/env Rscript
library(XXXX)
data=read.table("XXXX")
......
```
2. 使用 Rscript 這個指令去執行R code即可,國網電腦上面有已經裝好不同版本的Rscript程式,路徑如下:
```shell=
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v3.6.3/bin/Rscript
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v3.6.3_gcc_8.3.0/bin/Rscript
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v3.6.3_gcc_8.3.0.old/bin/Rscript
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v3.6.3_gcc_v7.5.0/bin/Rscript
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v3.6.3_rstudio/bin/Rscript
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v4.1.0/bin/Rscript
```
3. 執行rscript 之指令: **請放到[工作指令稿](https://man.twcc.ai/cV1vwW9GRTql6ODnTREPhw?both#%E5%B7%A5%E4%BD%9C%E6%8C%87%E4%BB%A4%E7%A8%BF)裡面執行**:
```shell
/work/opt/ohpc/Taiwania3/pkg/biology/R/R_v4.1.0/bin/Rscript myRcode.r
```
4. 如果執行時有短少的 R package 請來信 bio.nchc@niar.org.tw,我們會協助您安裝。
### 執行 IGV
使用X11支援Terminal如mobaxterm即可display回用戶端做使用。
* Windows 參見: https://man.twcc.ai/cV1vwW9GRTql6ODnTREPhw?both#Mobaxterm%EF%BC%88for-Windows%EF%BC%89
* Mac 參見:
https://man.twcc.ai/cV1vwW9GRTql6ODnTREPhw?both#XQuartz%EF%BC%88for-Mac%EF%BC%89
**範例:**
```
/work/opt/ohpc/Taiwania3/pkg/biology/IGV/IGV_v2.10.3/igv.sh
```
### 如何使用 RAPIDS
#### Server端
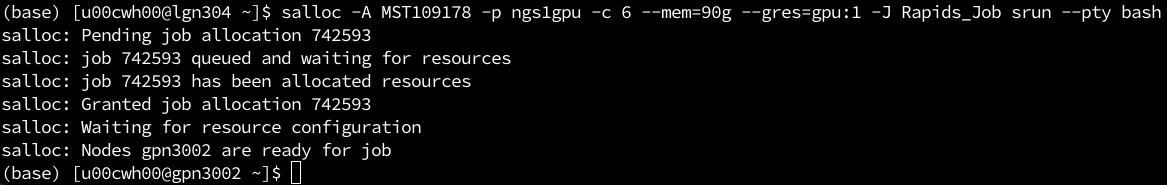
1. Allocate GPU partition
```wrap
salloc -A MST109178 -p ngs1gpu -c 6 --mem=90g --gres=gpu:1 -J Rapids_Job srun --pty bash
```
* 成功後出現下面訊息,記住node名稱與jobid,下面範例中allocate的node是gpn3002,jobid為742593
2. 設定環境變數
```
export SINGULARITYENV_TINI_SUBREAPER=1
ml singularity
```
3. 載入 RAPIDS singularity
```wrap
singularity run --nv \
/work/opt/ohpc/Taiwania3/pkg/biology/Rapids/rapids_cuda11.0/rapidsai_21.10-cuda11.0-runtime-ubuntu18.04.sif
```
4. 出現下面訊息後記住port,在下面範例中port是8888

#### 客戶端
5. 客戶端建立 ssh tunnel,{}中填入對應的資訊,就可以使用jupyter notebook 做訓練了。
`ssh -NfL {localport}:{nodename}:{port} {username}@t3-c4.nchc.org.tw`
範例:
```
ssh -NfL 10002:gpn3002:8888 u00cwh00@t3-c4.nchc.org.tw
```
6. 使用瀏覽器打開
網址:localhost:10002
7. 範例位置在 /rapids/notebooks/
可以cp到自己家目錄執行
8. 使用完成後,請記得刪除保留的GPU資源,以免資源浪費與產生額外的費用
`scancel -i $jobid`
範例:
```
scancel -i 742593
```
### 如何使用 Parabricks
請參考下列使用說明連結:
[T3 生醫節點 Parabricks 使用說明](https://man.twcc.ai/@nchcbio/ryS4mnjZh "Parabricks")
### 如何使用 AlphaFold v2
請參考下列使用說明連結:
[台灣杉三號生醫服務平台AlphaFold2說明](https://man.twcc.ai/@nchcbio/HkHhchdpo "AlphaFold2中文")
[Manual on Using AlphaFold2 on Taiwania3 Bio](https://man.twcc.ai/@nchcbio/HJOE34NCs "AlphaFold2")
### 如何使用Google Drive、Amazon等雲端資料資料
1. 先載入 Rclone
```
module load biolgy
module Rclone/1.59.0-DEV
```
2. 點選下面說明網頁中平台,根據說明進行設定
https://rclone.org/docs/
## 修訂紀錄
| 修訂日期 | 變更說明 |
| -------- | -------- |
|2025/05/13| 變更客服人員email |
|2025/06/10| 舊版軟體風險告知 |
|2025/06/12| 硬碟資料刪除政策 |
##
## Last updated: 2025/06/12 09:14